10th European Meeting on
HIV & Hepatitis – Treatment Strategies & Antiviral drug resistance –
Barcelona, 28.-30.03.2012
 Neue
Themen, neue Methoden und neue Erkenntnisse
Neue
Themen, neue Methoden und neue Erkenntnisse

Wendeltreppe im Turm der Kirche Sagrada
Familia in Barcelona
Vor allem im Bereich der HIV Epidemiologie gehörte natürlich die medikamentöse Transmissionsprophylaxe zu den Hauptthemen. Die verschiedenen Strategien, wie PrEP in Form von systemisch wirksamem Tenofovir/Emtricitabin, lokal wirkendem vaginalen Tenofovir-Gel oder als grundsätzliche Behandlung jedes Infizierten, wurden von Carlo Federico Perno, Tor Vergata University Hospital, Rome, vorgestellt (Einführungsvortrag, Session 1). Aufgrund der Datenlage ist eine abschließende Beurteilung der Relevanz von Virusresistenzen gegen Tenofovir in dieser Darreichung als Vaginalgel derzeit nicht möglich. Die in der Caprisa 004-Studie beobachteten hohen Spiegel bei vaginaler Applikation von Tenofovir-Gel können möglicherweise selbst bei geringgradiger Virus-Resistenz noch Schutz bieten, wobei diese Mutationen, falls es trotzdem zu einer Infektion kommt, selbst zu keiner Erhöhung der Resistenz gegen Tenofovir zu führen scheinen.
Resistenz-Überwachung bei PrEP
In diesem Zusammenhang kommt der Überwachung übertragener Resistenzen eine hohe Bedeutung zu. Solange nur wenige für eine Tenofovir-Resistenz relevante Mutationen in den übertragenen Virusstämmen beobachten werden, wie für Deutschland im Rahmen der Serokonverter-Studie des Robert Koch-Institutes gezeigt (Meixenberger, O_02) (Abb. 1), ist eine Einschränkung der Wirksamkeit der verschiedenen Strategien nicht zu erwarten. Da aber Tenofovir derzeit eines der am häufigsten eingesetzten Medikamente im Rahmen der HIV-Therapie ist, besteht auch hier das Risiko, dass Resistenz-assoziierte Mutationen in den übertragenen Virusstämmen weiter zunehmen werden. Die phylogenetische Analyse der HIV Epidemie in Dänemark (Audelin, O_04) verdeutlicht hierbei mehrere Aspekte: So bilden länger infizierte Patienten nur selten den Ausgangspunkt für ein neues Infektionscluster; außerdem sind solche Cluster meistens klein. Als hauptsächliche Infektionsquelle werden in dieser Studie jene Infizierten genannt, die erst seit kurzer Zeit infiziert sind, und bei denen der Infektionsstatus möglicherweise noch gar nicht bekannt ist. Die hieraus entstehenden Infektionscluster sind deutlich größer. Während also in dieser Studie epidemiologisch eine Trennung zwischen bereits Behandelten und frisch Infizierten beobachtet wird, was das Risiko einer Resistenzübertragung in der Gesamtpopulation eher senken würde, kann sich ein etabliertes resistentes Virus aber auch im Gesamtkollektiv schnell ausbreiten. Eine niederländische Arbeitsgruppe zeigte dazu (Hofstra, O_03), dass solche resistenten Viren über mehrere Stufen stabil weiter übertragen werden können.
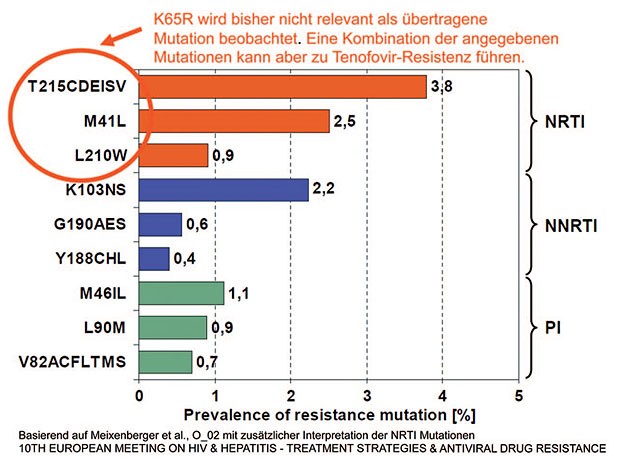
Abb. 1 Prävalenz von Resistenzmutationen
Neue Methoden
Ein weiterer ausführlich diskutierter Themenblock waren die neuen Untersuchungstechniken mittels „Next-Generation-Sequencing“. Zwar ist der Einsatz dieser Verfahren noch hauptsächlich auf Studien beschränkt, aber eine klinische Anwendung in der Diagnostik ist bereits durchaus absehbar. Aufgrund eines immer präziseren Nachweises selbst von minoren Viruspopulationen nimmt auch die Bedeutung der Fehlerraten des jeweils verwendeten Systems zu. Die schon jetzt hohe Bedeutung der Bioinformatik wird deshalb weiter zunehmen, vor allem wegen der enorm großen Datenmengen, die verarbeitet werden müssen. Inzwischen stehen nun die benötigten Werkzeuge, zumindest in Form von Prototypen, zur Verfügung (Rodriguez, Break-out session 6A). Kaum dass sich der Zuhörer an Systeme wie „454“ oder „Genome Sequencer (GS)“ gewöhnt hat, gibt es inzwischen neuere Geräteplattformen wie „IonTorrent“ oder „miSeq“, die sich im Forschungsbereich bereits bewähren (Thielen, O_21). Die Zukunft holt uns hier womöglich schneller ein als vor wenigen Jahren gedacht. So werden von „Oxford nanopore technologies“ bereits Sequenziergeräte in der Größe eines USB-Sticks entwickelt.
Low-Level-Viremia
Ein weiteres Thema, das auf Jahre Beschäftigung verspricht ist, die „low-level-viremia“. Allein die Definition macht schon große Probleme. Während einzelne Arbeitsgruppen darunter hauptsächlich Viruslasten unterhalb der häufig verwendeten Nachweisgrenze von 50 Kopien/mL verstehen, definieren andere Arbeitsgruppen den Bereich deutlich großzügiger und beziehen z.B. persistierende Viruslasten bis 200 Kopien/mL mit ein. Zumindest anhand einer Gruppe von 10 Patienten konnte gezeigt werden, dass eine persistierende Viruslast in diesem Bereich nicht zu Resistenz führen muss (Ehret, P_51). Verkompliziert wird die Situation durch die eingeschränkte Reproduzierbarkeit der Ergebnisse bei Viruslasten im sehr niedrigen Bereichen (<50 K/mL). Hier spielen statistische Effekte eine sehr ausgeprägte Rolle (Wensing, Break-out session 5A). In diesem Zusammenhang wurde auch wieder betont, die Viruslast in anderen Körper-Kompartments, wie z.B. dem ZNS, zu bestimmen und ggf. die Resistenzbestimmung auch von dort anzustreben, da hier Unterschiede existieren können.
Provirale DNA

Österreichisch-Schweiz-Deutsche Expertengruppe * J. Berg – AKH Linz, S. Christensen – CIM Münster, M. Däumer – IIG Kaiserslautern, R. Ehret – mib Berlin, M. Fraune – Universität Köln, K. Grabmeier – Universität Wien, B. Haas – LKH Graz, R. Kaiser - Universität Köln, T. Klimkait – Universität Basel, E. Knops - Universität Köln, N. Lübke - Universität Köln, M. Obermeier – mib Berlin, C. Paar – AKH Linz, B. Payer – Universität Wien, A. Pironti – MPI Saarbrücken, A. Sikorska – Universität Köln, H. Stoiber – Universität Innsbruck, A. Thielen – IIG Kaiserslautern, J. Verheyen – Universität Köln, H. Walter – Universität Erlangen
Ein immer wieder diskutierter Ansatz ist die Bestimmung der zellulären HIV-Last aus proviraler DNA in der Hoffnung, hier zusätzliche Informationen zu gewinnen. Die Daten hierzu, z.B. aus der MONET-Studie, erscheinen auf den ersten Blick nicht vielversprechend (Geretti, O_06). So konnte zwischen der Gruppe von Patienten auf DRV/r-Mono-Therapie gegenüber DRV/r + 2 NRTI zu den Zeitpunkten Woche 48, 96 und 144 im Median kein signifikanter Unterschied in der proviralen Viruslast im Vergleich zur Baseline (Therapiebeginn) festgestellt werden. In der Untergruppe dieser Studie mit Patienten, bei denen mindestens einmalig der Nachweis einer Viruslast von mehr als 50 Kopien/mL gelang, zeigte sich dann doch ein signifikanter Unterschied in der proviralen Viruslast zu solchen Patienten, bei denen die Viruslast nie über 50 Kopien gelegen hatte. Letztendlich kann die Kernfrage der niedrig nachweisbaren Viruslasten, ob es sich um „echte“ Replikation oder nur eine Ausschwemmung von Virus aus reaktivierten Zellen handelt, mit den derzeit verfügbaren Verfahren nicht beantwortet werden.
Tropismus-Test
Ein weiteres viel diskutiertes Thema bleibt die genotypische Tropismustestung. Die von Richard Harrigan1 vorgeschlagene Bestimmung des Tropismus als 3-fache Parallelbestimmung („Triplikat-Testung“) sollte vor allem eine größere Befund-Sicherheit für die Patienten bringen. Die Übereinstimmung der Ergebnisse eines Triplikats mit den nur einfach durchgeführten Bestimmungen ist jedoch sowohl für virale RNA, als auch für provirale DNA ausreichend groß (de Luca, O_17).
Während in Europa und Nordamerika hauptsächlich Patienten mit HIV-1 Subtyp B Virus behandelt werden, sind in anderen Teilen der Welt non-B Subtypen von HIV-1 häufiger und nehmen inzwischen durch Migration auch in Europa deutlich zu. Viele der Werkzeuge, die zu Diagnostik und Monitoring verwendet werden, sind aber auf dem HIV-1 Subtyp B entwickelt worden und somit besonders an Subtyp B angepasst oder wie z.B. geno2pheno[coreceptor], sie sind gar auf den entsprechenden Datensätzen trainiert. Dass der Prozentsatz X4-troper Viren in einer HIV-Population mit überwiegend non-B Viren sich deutlich von dem in Subtyp B-dominierten Patientenkohorten unterscheidet, wurde bereits beschrieben (u.a. Obermeier, P_68, To P_02). Im Vergleich zu einem phänotypischen Verfahren konnte hier eine Diskrepanz mit geno2pheno vor allem bei den HIV-1 Subtypen CRF01_AE und D gezeigt werden. Ein Therapie-Risiko für die Patienten besteht in diesen Fällen vermutlich nicht, da geno2pheno[core-ceptor] die Virusstämme fälschlicherweise als X4-trop deklariert, so dass die Patienten von Maraviroc-Therapie eher ausgeschlossen werden. Die daraus entstehende Einschränkung der Therapieoptionen kann aber auch problematisch sein.
HCV-Resistenz
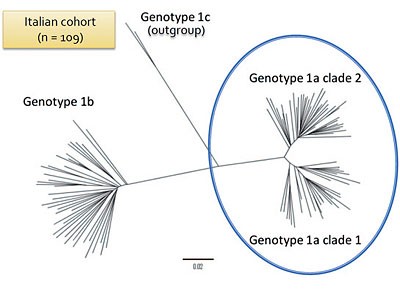
Abb. 2 HCV Genotyp 1a NS3-Sequenz zeigt zwei unterschiedliche Stämme. Nach Vicenti, JAC 2012
Während für die neuen HCV-Medikamente das Gros der Resistenz-assoziierten Mutationen bereits durch die Zulassungsstudien bekannt ist, ist ihre klinische Bedeutung weiterhin unklar (Forns, Einführungsvortrag für Session 3). Wahrscheinlich kann dies erst mit den Ergebnissen der kommenden Interferon-freien Therapieansätze beurteilt werden. Das dramatisch unterschiedliche Ansprechen der verschiedenen Genotypen auf die klassische HCV-Therapie ist hinlänglich bekannt. Die derzeit zugelassenen neuen HCV-Therapien sind nur für Genotyp 1 ausreichend wirksam, wobei sich vor allem bei der Resistenzentwicklung noch ein deutlicher Unterschied zwischen Genotyp 1a und Genotyp 1b zeigt. Daher erscheint vor allem Genotyp 1a problematisch, und natürliche Polymorphismen treten viel häufiger auf, die mit Resistenz assoziiert sein können (Di Maio, O_11, Plaza, O_12, De Luca, O_14). Verkompliziert wird dies für Genotyp 1a außerdem durch die Tatsache, dass er in zwei distinkten Untergruppen existiert, die möglicherweise auch unterschiedliche Resistenzcharakteristiken zeigen (Abb. 2).
Fazit
Trotz seiner wichtigen Öffnung in Richtung Hepatitis behält der Kongress seinen Schwerpunkt im Bereich HIV. Dies wird sich, solange die klinische Relevanz der Resistenzen gegenüber den neuen Medikamenten in der HCV-Therapie noch nicht belegt wurde, auch in den nächsten Jahren nicht ändern. Im Bereich HIV werden uns Themen wie (persistierende) niedrige Viruslasten und die entsprechende Diagnostik noch weiter beschäftigen, wobei die absehbare Zulassung der nächsten Integrase-Inhibitoren (Elvitegravir, Dolutegravir) Themen wie Therapieplanung und Resistenz wieder in den Vordergrund bringen könnte.
1 McGovern, R. A. et al. Population-based V3 genotypic tropism assay: a retrospective analysis using screening samples from the A4001029 and MOTIVATE studies. AIDS 24, 2517–2525 (2010)









 Diese Seite weiter empfehlen
Diese Seite weiter empfehlen